最終更新日 2023年3月10日 by Quark Staff
Chem Expand Bitは、SMILES等の化学構造データから、フィンガープリントを使ってビット表現へ変換し、さらに機械学習へ渡すためにテーブルカラムへ展開します。これにより、構造データをもとに、ランダムフォレストを使った物性予測や、クラスタリングによる教師なしでの構造分類が可能になります。
環境設定
※既に環境設定がお済みの場合は、読み飛ばして下さい。
Chemシリーズアプリをお使いいただくには、環境設定が必要です。KNIMEを起動し、追加のExtensionをインストールします。File>Install KNIME Extensionsをクリックします。
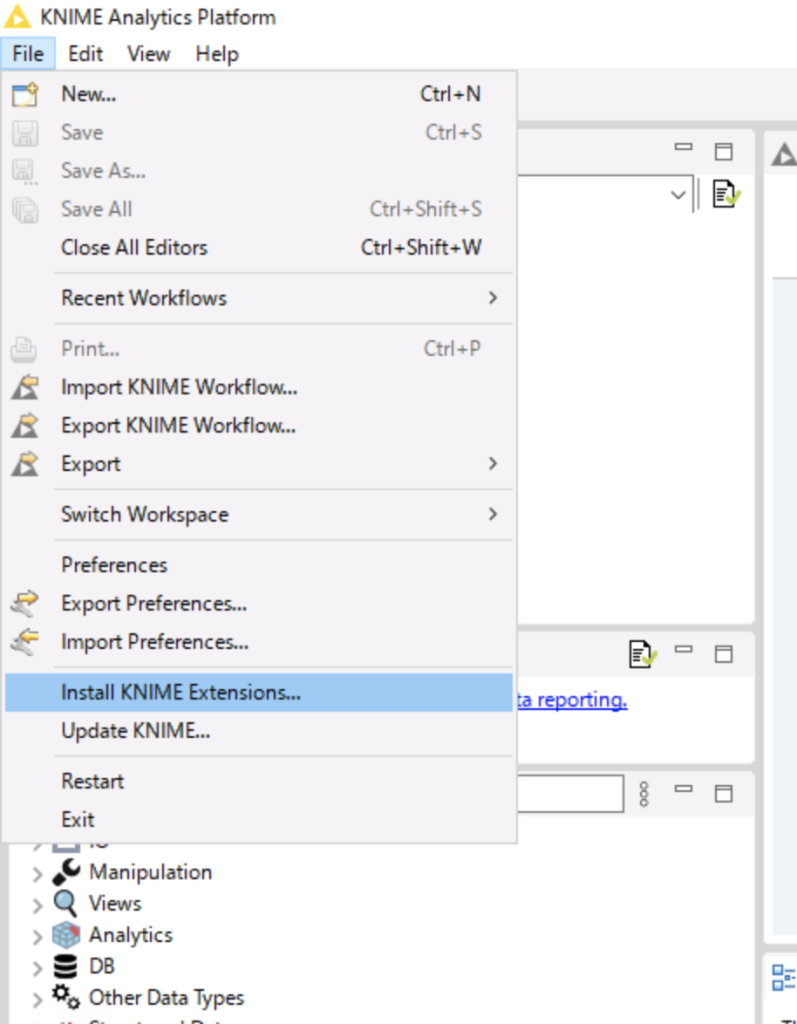
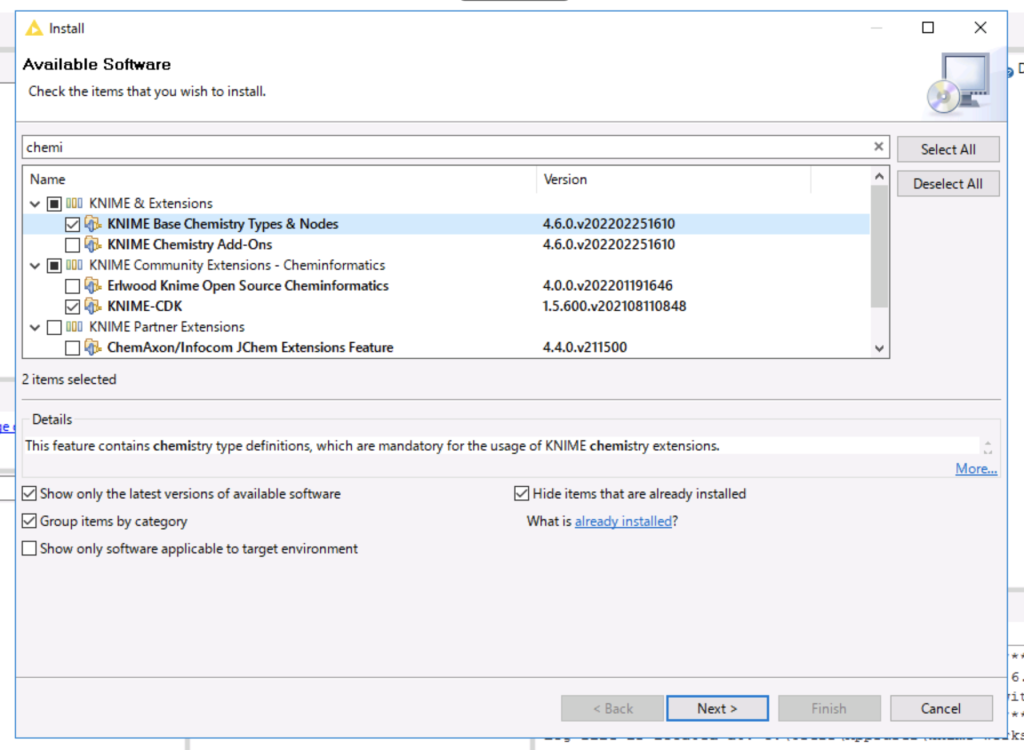
chemiで検索し、「KNIME Base Chemistry Types & Nodes」と「KNIME-CDK」を選択してインストールします。
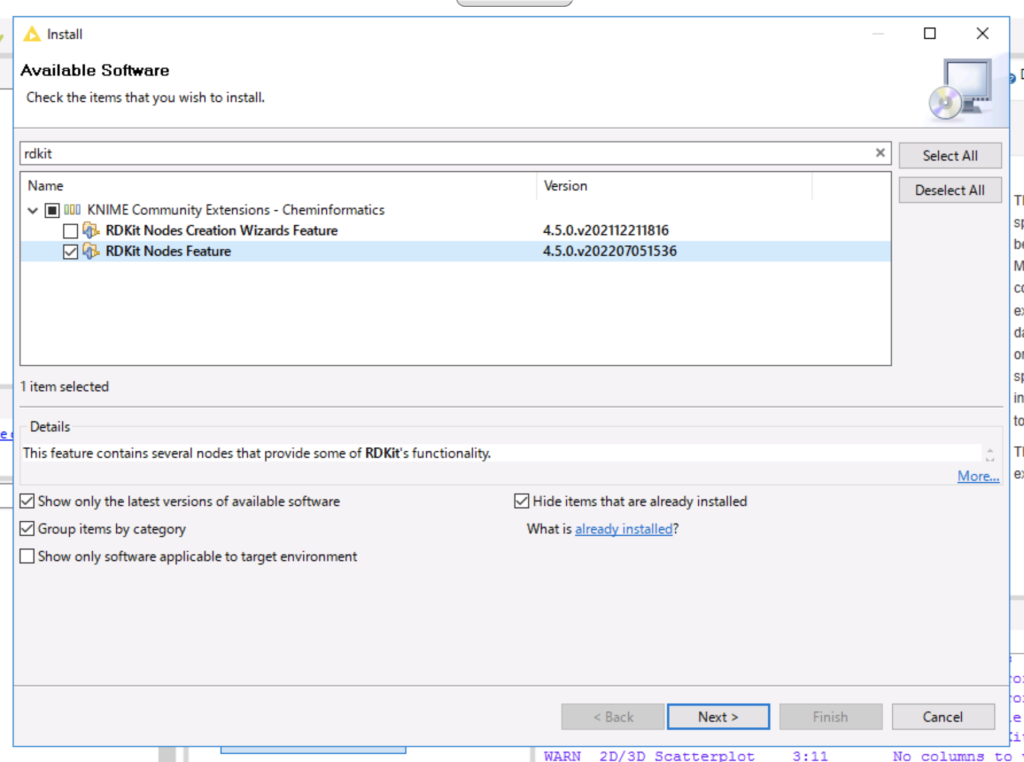
次にrdkitで検索し、「RDKit Nodes Feature」を選択してインストールします。
インストール
次に、アプリをインストールします。Get AppsからChem Expand Bitをダウンロードし、解凍してからインストールして下さい。Quark Appsタブ右端のMy Appsに登録されます。
使い方
検索対象とするデータをロードします。構造データとしてSMILESを含んでいる必要があります。
Chem Expand Bitを起動します。
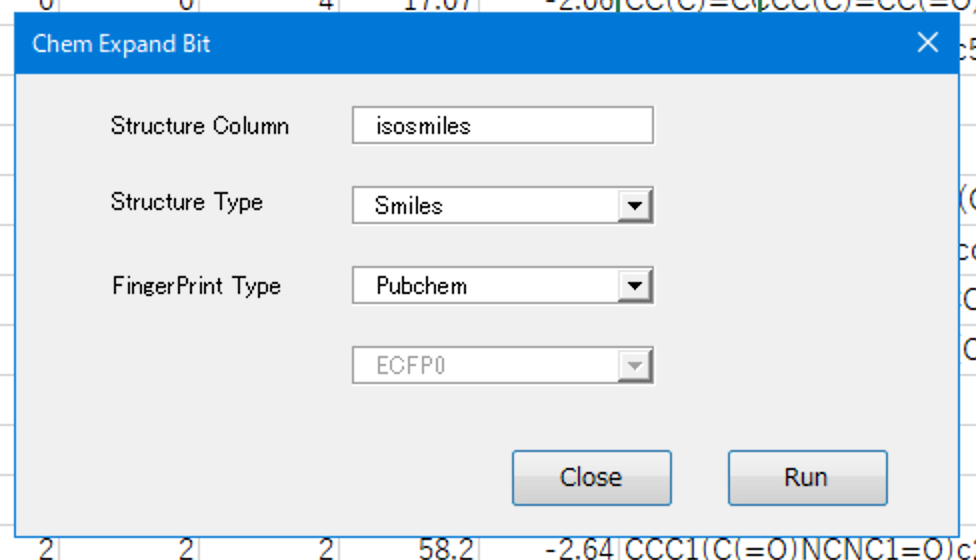
Structure Columnに構造データの列を指定し、Structure Typeでそのデータ型を選択します。そして所望のFingerPrint Type(デフォルトはPubchem)を選択し、準備ができたらRunします。
出力ファイル
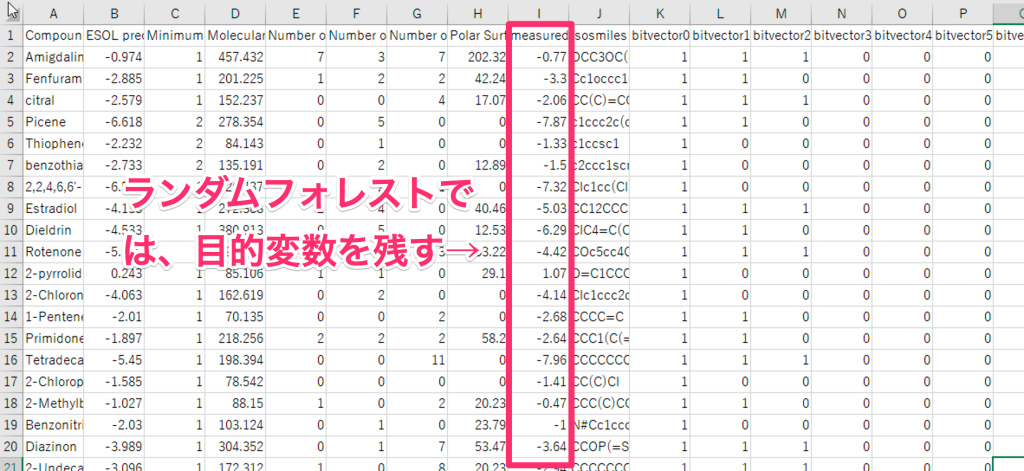
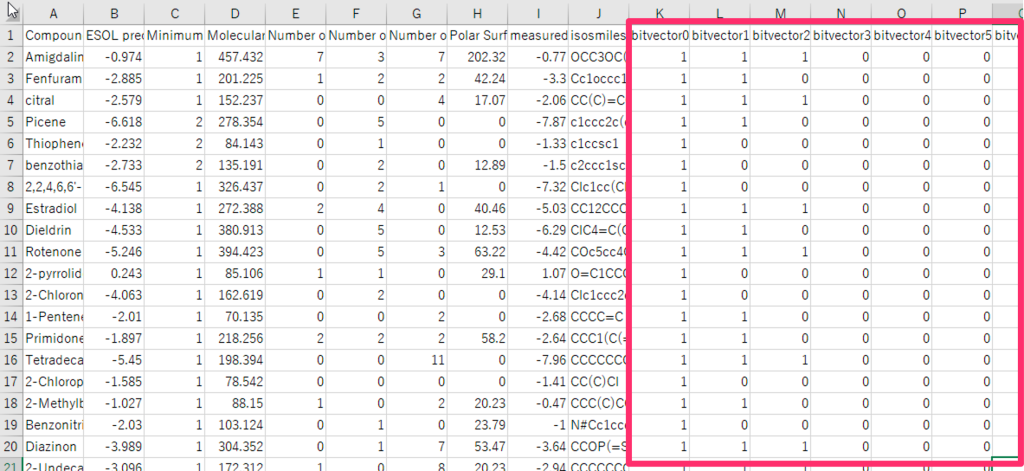
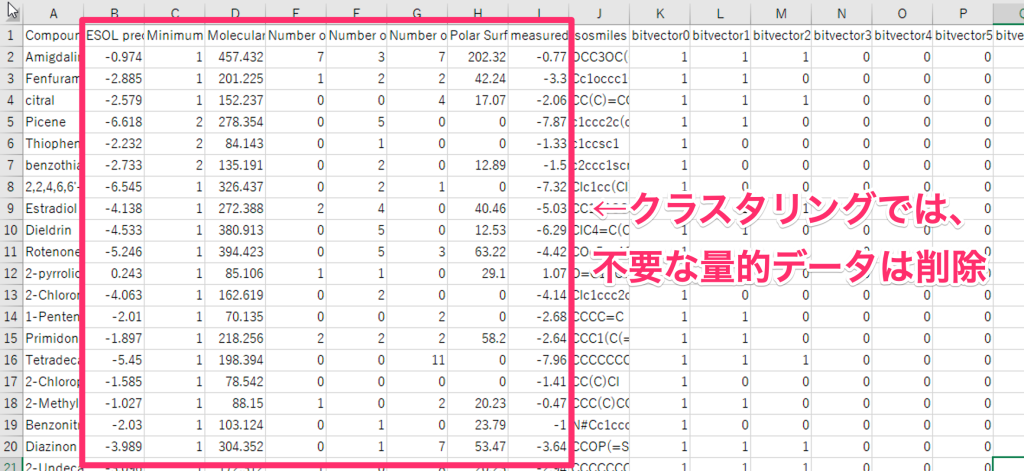
処理が完了すると、bitvector列が追加されます(下図赤枠)。このbitvector列が、機械学習へ渡したときの説明変数になります。
機械学習における注意点
例えば構造類似性で化合物を分類するには、上記bitvectorをもとにClusteringを実行します。その場合、不要な量的データは削除してから実行して下さい。
また、例えば構造類似性をもとに化合物の物性予測を行うには、上記bitvectorをもとにRandom Forestを実行します。その場合、bitvector列に加え、目的変数となる列(この例では溶解度の列)を残し、他の列は削除してから実行して下さい。